2025年10月24日,《自然—通讯》(Nature Communications)在线发表我院薛卫副教授BioAI实验室与资环学院沈其荣院士团队LorMe实验室的交叉学科研究成果《LorBin: Efficient binning of long-read metagenomes by multiscale adaptive clustering and evaluation》。该工作聚焦生物信息学底层算法,以三代宏基因组分箱为切入点,研发了多尺度自适应聚类与迭代评估算法LorBin,解决了宏基因组研究过程中重构单菌基因组数量少、质量差的共性难题。该研究积极响应了我院重组后关于将人工智能深度融入并赋能农业应用基础研究的重要部署,为我校推动AI赋能的前沿交叉学科建设与发展提供了有力支撑。

BioAI实验室长期从事AI与农业应用基础研究。近年来,BioAI实验室主动出击,积极对接沈其荣院士团队LorMe实验室,以农业废弃物、果蔬病害为研究对象,聚焦农业表型、生物大数据及深度学习模型研发等关键科学难题:一、构建全球首个深度学习腐熟度预测模型;二、提出梨树炭疽病早期检测算法;三、构建花粉、果实等表型图像增广、识别检测算法,相关研究发表在Bioresource Technol、Plant Phenomics、植物营养与肥料学报、南京农业大学学报等期刊。
以复杂环境微生物为对象,聚焦自然语言处理的长文本以及用户可及性的难题,一、研发了基于基因组全局性特征的深度交叉融合网络算法DCiPatho,用于复杂环境微生物群落中致病菌的精准识别;二、构建免费的微生物生态多组学在线分析平台CFViSA。相关研究发表在Brief Bioinform、Comput Biol Med等期刊。随后沈其荣院士提出宏基因组测序,特别是三代宏基因组是揭示农业微生物与全球大健康关系研究的手段,要集中精力攻关、解决一些共性难题。
深入调研发现,三代宏基因组研究面临着原始数据量庞大、碱基序列错误率高和分析算法不完善等卡脖子难题,尤其是在分箱重构高质量单菌基因组这一关键环节。宏基因组分箱是指将测序得到的碱基序列(reads)进行组装,形成更长的序列片段(contigs),随后将这些片段分配到若干个箱(bins)的过程。在理想情况下,每个箱对应一个微生物的基因组,即宏基因组组装基因组(MAGs:Metagenome-Assembled Genomes)。深入剖析后发现,这个过程看似简单,但挑战很大。既要克服复杂微生物群落中物种组成与丰度的不确定性、未知物种参考基因组缺失的限制,还需区分近缘物种、甚至菌株水平的遗传变异规律,更要解决原始数据利用率、长序列编码、特征提取、异形分布数据聚类与簇质量评估算法等卡点。

为攻克三代宏基因组分箱的难题,两个实验室共同组建攻关小组。该小组汇聚了来自瑞士、美国和巴基斯坦的海外合作伙伴,在原始数据遴选和方案优化上面给出了宝贵建议,上海凌恩生物科技有限公司生信工程师和微软中国的算法工程师分别在CPU/GPU服务器的配置和算法架构上给予了帮助和建议。值得一提的是我院2022级硕士研究生刘佐(已毕业),在获得录取通知书后第一时间进入高强度的工作状态,主要负责算法研究与实验任务,面对最前沿的AI技术,不畏艰难、深入钻研,首次参与高水平研究课题就取得重大突破,为我院青年学子树立了优秀榜样。资环博士生张耀中完成大量生信分析工作,在分析挖掘稀有物种,以及解析关键生物学现象方面发挥重要作用。
历经3年协同攻关,攻关小组创新了一种三代宏基因组长读分箱的无监督深度学习工具LorBin。该工具针对分箱全过程的卡点,从技术原理上提出四项针对性创新设计:1)适配变分自编码器训练学习复杂生境contigs特征分布,克服DNA大语言模型在处理长序列时面临的诸多挑战;2)构建两阶段多尺度自适应迭代聚类算法应对复杂物种空间分布,回收更多独特的未知微生物类群;3)在聚类过程中引入单拷贝基因集等信息引导的循环分箱质量评估模型,提升分箱质量和原始数据利用率;4)采用Transformer模型与统计概率模型,评估簇在高维空间的凹凸性对分箱效果的影响,解决高维生物数据嵌入特征在降维前后空间分布一致性评估的难题。
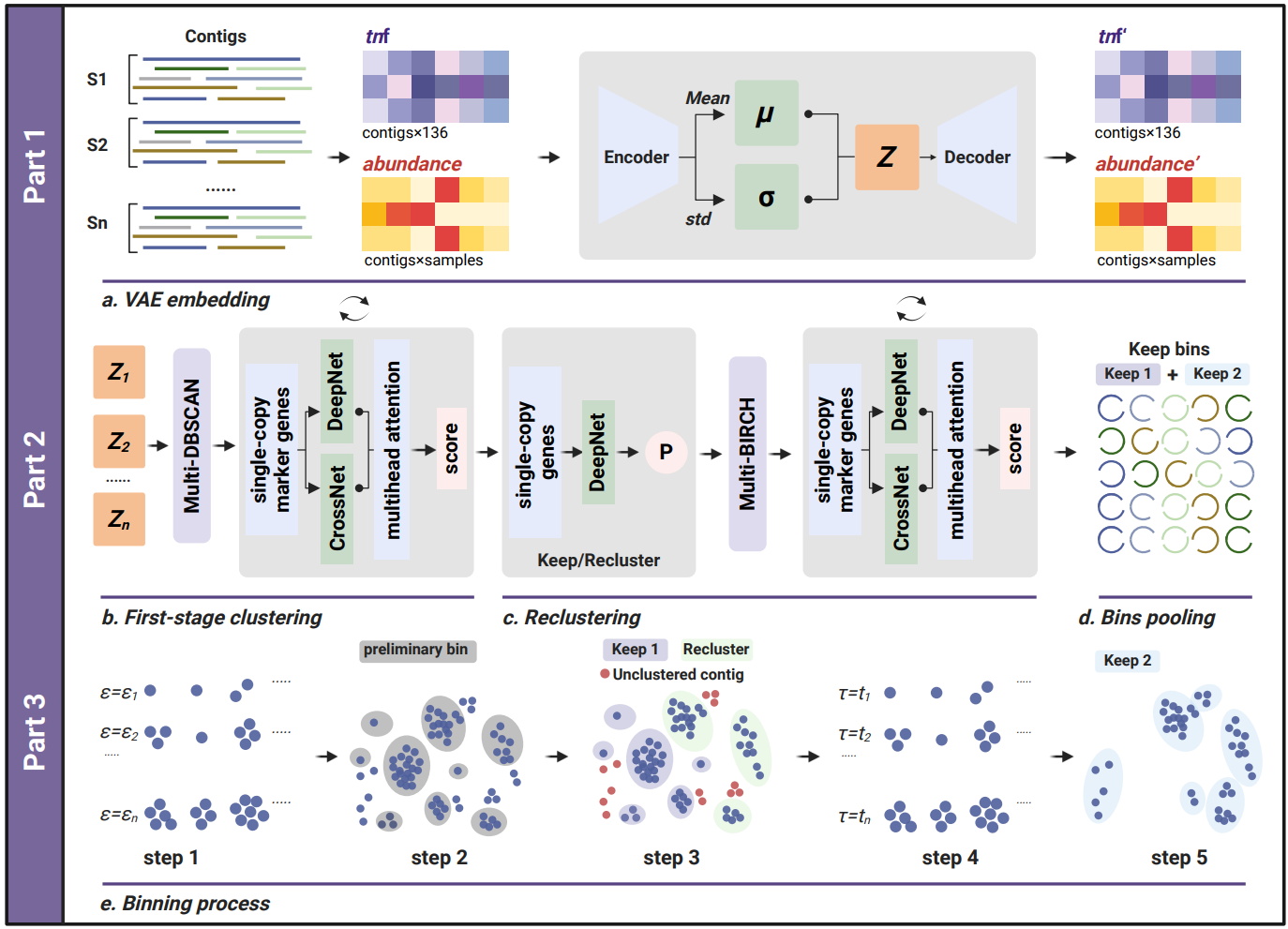
基准测试表明,LorBin的性能卓越,整体优于SemiBin2、VAMB和COMEBin等6种高性能深度学习算法。LorBin分箱的质量更高、捕获稀有物种的能力更强,重构的高质量MAGs比现有工具多15–189%,识别特有物种数量更是其他工具的2.4–17倍。该工具的运行效率高、可扩展性强、易用,在32核CPU、64GB内存和0.3*NVIDIA A800 GPU配置下,比SemiBin2和COMEBin等高性能工具快2.3–25.9倍。
LorBin的成功研发是产教学研的典型研究案例,更是AI与生物学的双向奔赴。这并非简单的技术嫁接,而是一次南农生物学与人工智能两大学科之间的深度融合。在这一过程中,BioAI实验室扮演着“算法建筑师”的角色,凭借强大的AI工具构建精妙的计算模型;而LorMe实验室则如同“微生物向导”,以其对微观世界的深刻理解,确保模型在持续迭代中不断逼近真实的生物学问题。这种双向奔赴最终构筑的创新生态圈,正是驱动未来科学发现的核心引擎。它为我院如何与其他学科交叉合作提供了极具参考价值的范本。未来,BioAI实验室将继续聚焦生物信息学重要课题,沿着“从微观机理到系统规律”的路径,向更广阔的生物学疆域开拓。
该研究得到国家自然科学基金(重大项目、杰出青年项目和面上项目)和中央高校基本业务费等项目的资助。yl88858cc永利官网薛卫副教授、研究生刘佐(已毕业)和资环学院博士生张耀中为共同一作者,资环学院韦中教授和江高飞副教授为共同通讯作者,资环学院Alexandre Jousset教授(国家外籍杰青获得者)、Waseem Raza副教授(现中国热带农业科学院热带生物技术研究所)、博士生李亚蓉、上海凌恩生物科技有限公司陶晔和钱俊博士以及贝勒医学院Fritz Sedlazeck副教授和微软中国高级工程师姜立等参与了该研究,沈其荣院士、赵方杰教授和徐阳春教授等共同指导了该研究。
全文链接:https://www.nature.com/articles/s41467-025-64916-8